

美国Molsoft公司明星产品综合的分子模拟研究平台ICM-Pro
关于Molsoft
Molsoft 公司成立于1994年,最初名为Biosoft,后于1995年更名为Molsoft.Molsoft公司的分子模拟技术以内部坐标力学的方法为基础,为生物结构学的各种任务和合理的药物设计提供了解决方案。
ICM项目启动于1985年,从此以后得到了持续的发展。 2000年, Abagyan博士的合作伙伴Maxim Totrov博士加入ICM成为ICM的首席科学家,2002年, Eugene Raush 作为高级软件工程师加入该团队。ICM拥有一个强大地管理研发和软件研发团队,充满活力和创造力的环境让Molsoft得以解决蛋白质组学和合理药物设计中最棘手的问题。
关于ICM-Pro
ICM-Pro可以使生物学家或化学家快速访问高品质的互动式3D视图的结构数据库。在短短的几秒钟内,用户可以浏览数百个感兴趣的化合物并加载它们的结构,可以对序列进行结构分析(保守区或突变),三维结构的构建,研究药物和蛋白或者是蛋白-蛋白的对接,可以研究表面的静电或者是疏水性。
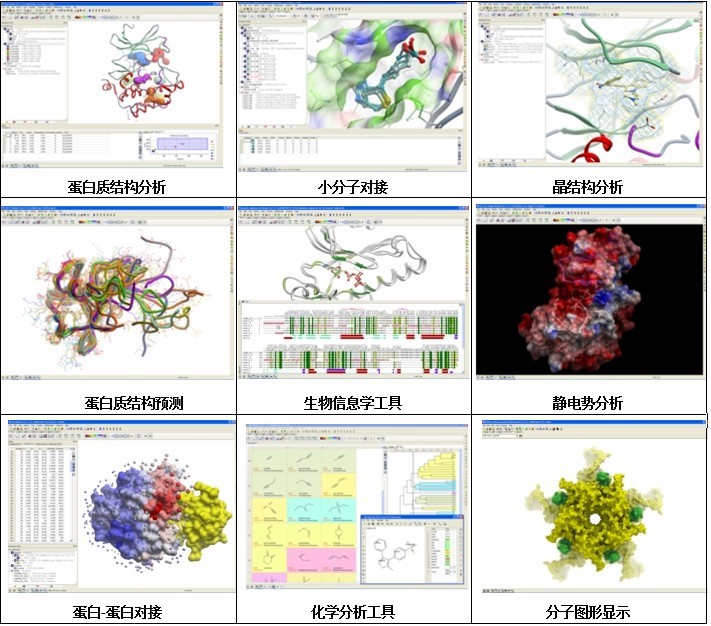
ICM-Docking—功能强大的柔性对接
ICM-Docking是快速准确的分子对接软件,支持小分子配体-蛋白、多肽-蛋白以及蛋白-蛋白的对接。同时该软件也支持快速准确的对接优化。人性化的交互图像显示画面和渐进式的使用说明使得软件的操作更加简单快捷。
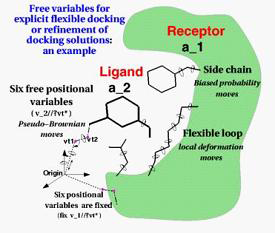
可以支持定义自变量的柔性对接,也可以对对接方案进行限定,如图所示:可以考虑受体侧链的柔性,不规则区域柔性。同时可以有六个可以自由变换的位置变量,或者是将此六个变量完全锁定。
功能
自动分子处理
在进行柔性对接之前,ICM-Docking可以对小分子配体进行准备工作,包括在局域拓扑连接的基础上分配MMFF原子类型,为配体加氢,分配电荷以及分子中可旋转键的识别。同时,ICM-Docking支持分子构象2D,3D之间的相互转换。
可视化格点电势
在柔性对接对接位点的确认时采用可视化的格点电势,使得对受体对接位点的确认更加方便快捷。
小分子配体对接
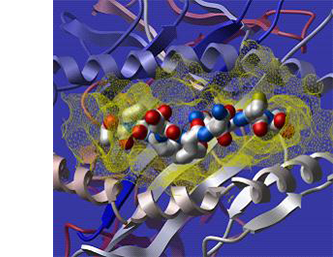 ICM-Docking提供了一系列独特的工具来模拟配体和蛋白之间的相互作用,利用格点的相互作用电势灵活的将全柔性的配体分子快速准确的对接进入受体蛋白分子中,同时允许用户任意指定全原子显示的蛋白受体某一侧链的柔性。
ICM-Docking提供了一系列独特的工具来模拟配体和蛋白之间的相互作用,利用格点的相互作用电势灵活的将全柔性的配体分子快速准确的对接进入受体蛋白分子中,同时允许用户任意指定全原子显示的蛋白受体某一侧链的柔性。
小分子配体的对接属于多构象对接,在对接过程中,ICM-Docking使用了随机的全局优化的方法产生配体的多构象,该过程将伪布朗的位置/扭转角步骤和快速的局部梯度优化方法联用。在优化算法中使用了连续可导的格点电势以达到局部优化的尽快收敛。ICM的对接算法可以跟踪分析优化轨迹以避免陷入次优构象,同时允许客户进行高效的分子构象空间搜索。对接配体所在的蛋白结合口袋可以添加表面,同时可将蛋白或受体的相关特性投射到所添加的表面上。
蛋白-蛋白对接
ICM-Dock包含了一个得到广泛验证的非常成功的算法,该算法不仅可以进行蛋白-蛋白对接,还可以预测蛋白之间相互作用的位点。在进行蛋白-蛋白对接时利用格点电势进行快速刚性的全局搜索,使用柔性侧链进行诱导契合以得到最优的对接构象。使用ICM优化对接区域方法预测蛋白作用位点,并用颜色标注蛋白-蛋白相互作用的热区,使用交互式表单针对每个氨基酸残基的ODA进行预测。如果没有预测的蛋白对接位点的话可以直接在整个蛋白表面对接。对接结果包含范德华作用,氢键作用,疏水作用,极性溶剂化能,脂性溶剂化能以及相关加权因子等。
相关案例:
1、ICM曾被用于从头对接一个溶菌酶到相应的抗体上,精度达到1.6Å(和晶体结构中配体构象的RMSD值) (Nature Struc.Biol., 1994, 1,259)。
2、Maxim Totrov and Ruben Abagyan 使用ICM的伪布朗对接和ICM侧链柔性限定等方法准确预测了β-内酰胺酶和蛋白抑制剂的结合方式(Nature Struc.Biol., 1996,3,290)。
多肽-蛋白对接
ICM-Dock除了配体-蛋白对接和蛋白-蛋白对接以外,还可以进行蛋白和柔性多肽的对接,此时对接算法使用了ICM的OBMCM全局优化的方法,并且使用了格点电势进行快速刚性的全局搜索。该对接过程和蛋白-蛋白对接相似,都属于柔性对接,使用诱导契合的方法以得到最优的对接构象。

相关案例:
1、含有SH2和PTB结构域的配体和蛋白的对接(Folding & Design, 1998, 3, 513)
2、霍乱毒素衍生肽和抗体的无限制柔性对接(Structure, 1999, 7, 663)
操作
有详细的分步操作说明,针对每一步操作都有详细的指导。用户可以根据操作说明自行学习。
采用图形化界面,操作方便。具有多种动态视觉效果,如动态阴影、立体切割等,以及多种动画控制演示。
参数修改
允许用户自行修改对接脚本,(该脚本用直观的ICM分子模拟脚本语言编写),以最大程度的满足客户的体系要求。
限制性对接,配体分子的部分基团可以被自动处理,限定在受体的预定义的对接位点附近。
核心优势
诱导契合:对接的配体,受体蛋白甚至是短的多肽都可以有柔性构象,在对接时相互调整,诱导契合对接,以获得最优化对接构象。
高效,快速。ICM对接支持配体分子的多构象对接和多分子的同时对接(一次100个)。
特殊的蒙特卡洛算法可以识别外消旋异构体
可在集群的不同机器上运行多个作业
硬件需求
100Mb的磁盘空间,1Gb或者更多的RAM(缓存空间)
256MB以上的显卡,推荐NVIDIA 512MB以上的显卡
支持系统
Windows 7/Vista/XP/NT/2000,
Linux/i386/AMD64,
SGI IRIX,
Mac OS X